FINPROTAX - Suomen hyönteisten tunnistus DNA-sekvenssien avulla
Tällä sivulla on lisätietoja PROTAX-sovelluksesta, jonka avulla voi tunnistaa Suomen hyönteisiä DNA-sekvenssien perusteella.
Tausta
Sovellus hyödyntää kansallista FinBOL-sekvenssitietokantaa. Tunnistuksessa käytetään menetelmää, joka antaa todennäköisyyden mihin taksoniin käyttäjän antama sekvenssi kuuluu taksonomian eri tasoilla (luokka, kunta, heimo, alaheimo, tribus, suku, laji). Tunnistuksen tuloksena saadaan myös todennäköisyys, jolla sekvenssi kuuluu ennestään tuntemattomaan lajiin tai muun taksonomiatason yksikköön. Tämä tulee tulkita tarkoittavan sellaista taksonia, jolle ei löydy sekvenssiä FinBOL-tietokannasta. Tähän ryhmään kuuluu mukaan myös ne tunnetut taksonit jotka ovat mukana nykyisessä taksonomiassa mutta joille ei löydy sekvenssejä nykyisestä FinBOL-tietokannasta.
FinBOL-viivakoodikirjaston kattavuus ilmenee lähdeartikkelista (Roslin et al. 2021). Koska luokitin perustuu tarkasti määriteltyyn geenialueeseen (viivakoodiin), käyttäjän tulee tietää millä PCR-alukkeilla hänen antamansa sekvenssi on monistettu.
Miten tulkita tunnistustuloksia
Käyttäjä saa tunnistustiedot tekstitiedostona. Yhdellä rivillä on sekvenssin luokitustulokset yhteen taksoniin siihen liittyvän todennäköisyyden kanssa. Tästä johtuen yhdenkin sekvenssin tulokset jakaantuvat yleensä usealle riville. Riippuen käyttäjän antamasta todennäköisyys-kynnysarvosta, samallekin taksonomiatasolle voi tulla useita tunnistuksia yhdeltä sekvenssiltä. Kullakin rivillä on viisi saraketta:
sekvenssiID taksonomia_taso finbif_koodi taksoni todennäköisyys
SekvenssiID tulee käyttäjän antaman FASTA-tiedoston header-rivistä.
Taksonomiassa on seitsemän tasoa: 1) luokka, 2) kunta, 3) heimo, 4) alaheimo, 5) tribus, 6) suku ja 7) laji. Taksonomiset nimet noudattavat Suomen lajitietokannassa olevia nimiä. Todennäköisyys tarkoittaa todennäköisyyttä jolla käyttäjän antama sekvenssi edustaa rivillä mainittua taksonia.
Alla on kolme esimerkkiä erilaisista tunnistustuloksista. Lisätietoa käytetystä tunnistusmenetelmästä löytyy tämän dokumentin lopusta.
Esimerkki 1: Luotettava lajitason tunnistus
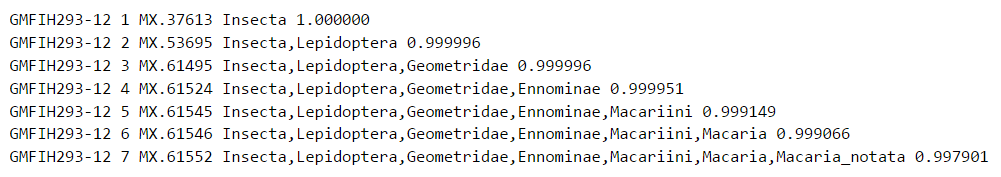
Todennäköisyydet pienentyvät aina kun mennään luokkatasolta lajitasoa kohti. Tässä esimerkissä todennäköisyys yhteen taksoniin on kuitenkin hyvin korkea jopa lajitasolla. Näinollen on hyvin todennäköistä että annettu sekvenssi ‘GMFIH293-12’ edustaa lajia Macaria notata.
Esimerkki 2: Luotettava tunnistus heimotasolle

Tässä esimerkissä tunnistus ulottuu alaheimo-tasolle, jolla suurimman todennäköisyyden saanut taksoni on nimetty tuntemattomaksi (unk lyhenteenä sanasta unknown). Se edustaa joukkoa taksoneita, joille ei ole sekvenssejä tietokannassa. Näihin kuuluvat sekä tunnetut että tuntemattomat taksonit. Esimerkin tunnistuksen tulkinta on että sekvenssi kuuluu todennäköisesti Braconidae-heimoon, mutta on sellaisesta Braconidaeen kuuluvasta alaheimosta, josta ei ole sekvenssejä nykyisessä FinBOL tietokannassa. Tähän kuuluvat sekvenssit, jotka edustaisivat Braconidaen tunnettuja alaheimoja että myös sekvenssit, jotka edustaisivat Braconidaen alaheimoja mutta joita ei vielä ole nykyisessä taksonomiassa mukana.
Huom.: unk-nimisille taksoneille ei ole FinBIF taksonikoodia, joten tulostiedoston 3. sarakkeessa on NA (Not Available).
Esimerkki 3: Sama viivakoodi eri lajeilla
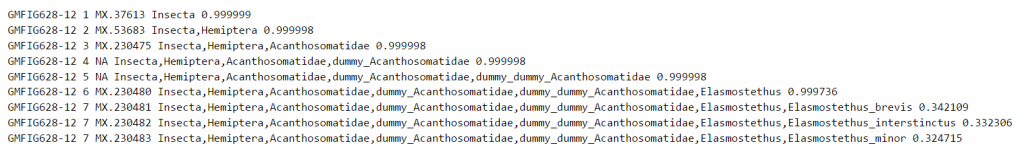
Tässä esimerkissä saadaan korkea todennäköisyys sukutasolle asti mutta sen jälkeen todennäköisyys jakaantuu tasaisesti kolmen lajin kesken. Syynä on se että COI sekvenssi ei ole informatiivinen erottelemaan näitä lajeja. Tunnistuksen tulkinta on, että sekvenssi edustaa hyvin todennäköisesti Elasmostethus-sukua ja on joko E. brevis, E. interstinctus tai E. minor. Tarvittaisiin lisätietoa jotta lajimääritys voitaisiin tehdä tarkemmin. Esimerkiksi jos tiedetään että joku näistä lajeista ei esiinny sillä paikalla josta sekvenssinäyte on kerätty, voidaan näiden kolmen vaihtoehtoisen lajin määrää pienentää.
Tässä esimerkissä tulee esille myös taksonien nimeäminen (joka ei liity yhteisen viivakoodin ongelmaan).
Tasoilla 4 ja 5 taksonien nimissä esiintyy sana "dummy". Tämä johtuu siitä että tunnistusmenetelmän käyttämässä taksonomiassa ei ole alaheimo- eikä tribus -nimiä Acanthosomatidae-heimon ja Elasmostethus-suvun välillä. Koska tunnistusmenetelmä edellyttää yhtenäisen polun olemassaoloa taksonomian juuresta lajitasolle asti niin että jokaisessa polussa on yhtä monta taksonomiatasoa, on puuttuvien tasojen nimet lisätty keinotekoisesti käyttämällä "dummy"-sanaa. Tämä nimeäminen ei vaikuta tunnistustuloksiin ja esimerkin tapauksessa sekvenssi siis kuuluu hyvin todennäköisesti Elasmostethus-sukuun.
Huom.: Koska "dummy"-taksoneille ei ole FinBIF koodia, tulostiedoston 3. sarakkeessa on NA.
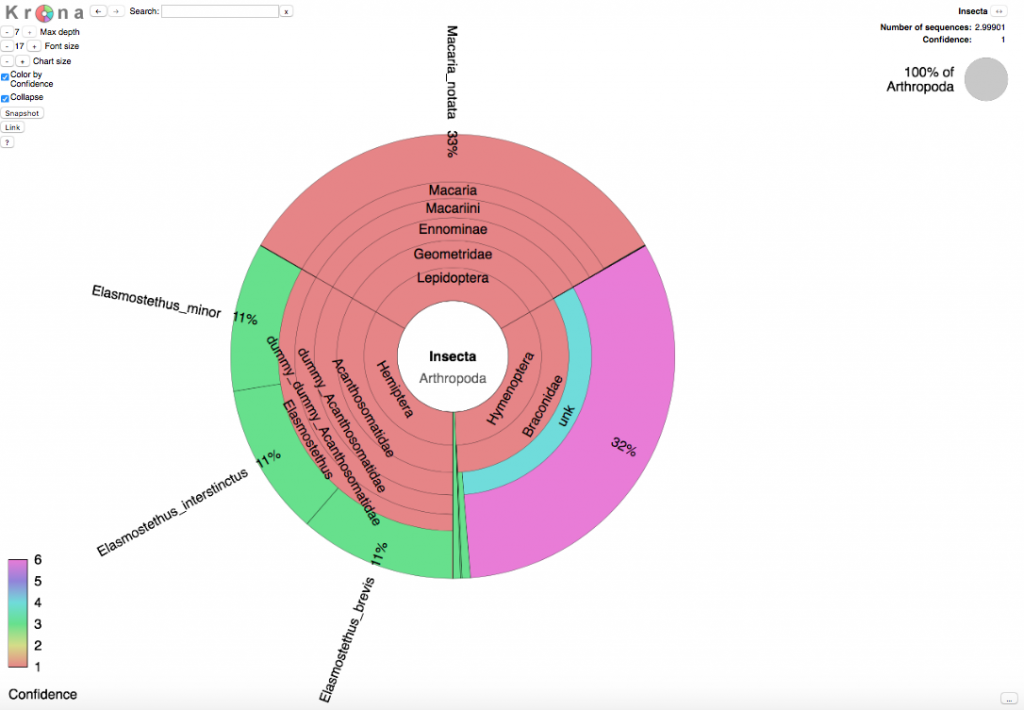
Taksonomian sektoridiagrammi
Tekstitiedoston lisäksi tunnistuksen tulokset saadaan myös visuaalisessa muodossa. Tekstitiedostossa on yksittäisten sekvenssien tunnistustulokset mutta visuaalisessa esityksessä kaikkien tunnistettujen sekvenssien tulokset esitetään yhtenä kuvana. Krona-ohjelmalla tehty kuva on interaktiivinen ja se tulee html-muodossa. Sen avulla voi tarkastella tunnistuksia taksonomian eri osissa kaikilla sen eri tasoilla.
Huom.: Krona-kuvassa esitetään vain ne taksonit, joihin on tullut todennäköisyys-kynnysarvon ylittäviä tunnistustuloksia.
Kun Krona-html kuvan vasemmassa yläkulmassa olevaa kohtaa 'Color by Confidence' klikkaa, jokainen taksoni esitetään kuudella eri värillä jotka edustavat eri tunnistuskategorioita. Nämä kertovat kuinka suuri osuus kyseiseen taksoniin tunnistetuista sekvensseistä on ylittänyt todennäköisyyden 0.9. Kategoriat 1-3 edustavat tunnistettuja taksoneita joille löytyy sekvenssi tietokanassa ja kategoriat 4-6 edustavat taksoneita joille ei ole sekvenssejä tietokannassa.
- (punainen). Yli puolet tunnistettujen sekvensseien todennäköisyyksistä on yli 0.9
- (keltainen). Vähintään yksi tunnistettu sekvenssi on saanut todennäköisyyden joka ylittää 0.9
- (vihreä). Yksikään tunnistettu sekvenssi ei ole saanut todennäköisyyttä joka ylittäisi arvon 0.9
- (turkoosi). Yli puolet tunnistetuista sekvensseistä on saanut todennäköisyyden yli 0.9
- (sininen). Vähintään yksi tunnistettu sekvenssi on saanut todennäköisyyden yli 0.9
- (purppura). Yksikään tunnistetuista sekvennseistä ei ole saanut todennäköisyyttä yli 0.9
Sektoridiagrammissa oleva taksonin koko vastaa siihen tunnistettujen sekvenssien määrän odotusarvoa. Tämä odotusarvo on taksoniin tunnistettujen sekvenssien todennäköisyyksien summa joka on laskettu ottaen mukaan kaikki käyttäjän antamat syötesekvenssit. Kaikki ne todennäköisyydet on huomioitu, jotka ovat ylittäneet käyttäjän antaman todennäköisyys-kynnysarvon. Allaoleva kuva näyttää esimerkin, jossa käyttäjä on antanut kolme edellisten esimerkkien sekvenssiä tunnistettavaksi.
Miten tunnistus toimii
Tunnistuksessa käytetään PROTAX-nimistä menetelmää. Se perustuu taksonomiaan, sekvenssitietokantaan sekä malliin joka muuttaa sekvenssien väliset samankaltaisuudet taksonien todennäköisyyksiksi. Kullekin käyttäjän antamalle sekvenssille (syötesekvenssi) tehdään seuraavat asiat:
- Käyttäjän antama sekvenssi linjataan piilo-Markov-mallilla (hidden Markov model) tietokannan sekvenssejä vastaan
- Lasketaan sekvenssietäisyydet (p-distance) syötesekvenssin ja kaikkien tietokannan sekvenssien välillä
- Sekvenssietäisyydet muunnetaan taksonien todennäköisyyksiksi tilastollisen mallin avulla (multinomiaalinen regressiomalli)
- Käyttäjälle annetaan lista niistä taksoneista, joiden todennäköisyydet ylittävät annetun kynnysarvon
Kahden sekvenssin välillä laskettava p-distance on määritelty eroavien nukleotidien lukumääräksi suhteessa sekvenssilinjauksen pituuteen. Ainoastaan ne nukleotidipositiot, joissa molemmissa sekvenssissä on joko A,C,G tai T lasketaan linjaukseen mukaan eli sekä aukot että ne poisitiot, joissa jommassakummassa sekvenssissä on N, jätetään laskennasta pois. Lisätietoja löytyy julkaisusta Roslin, et al. 2021. Lisätietoja PROTAX-menetelmästä löytyy julkaisuista Somervuo et al. 2016 a ja Somervuo et al. 2016b.
Huom.: Nykyinen FINPROTAX-versio tunnistaa ainoastaan luokkia Insecta ja Archnida
Tunnistin on opetettu antamaan taksonien todennäköisyyksiä Suomen hyönteisille. Suurin osa FinBOL-tietokannassa olevista sekvensseistä edustaa Insectaa ja muutama Arachnidaa. Tunnistimella ei ole minkäänlaista tietoa COI-sekvensseistä näiden luokkien ulkopuolelta. Näinollen jos käyttäjä antaaa syötesekvenssiksi jotain kyseisten luokkien ulkopuolelta, tunnistin ei pysty kertomaan mitä tällaiset sekvenssit edustavat. Suurimmassa osassa tapauksia tällaiset sekvenssit luokittuvat suurella todennäköisyydellä taksoniin "Insecta,unk" ja pienellä todennäköisyydellä johonkin tunnettuun tarkempaan taksoniin kunta-tasolla tai sen alapuolella.
Käytännön vinkki: Jos käyttäjä haluaa tunnistaa suuren määärän sekvenssejä, laskennan kannalta on edullisempaa jos näistä pystytään ensin suodattamaan pois jollain toisella luokittimella sekvenssit, jotka eivät kuulu Arthropodaan, jolloin jäljelle jää pienempi määrä sekvenssejä jotka kaikki kuuluvat siihen taksonomiaan, jota vastaan FinBOL-sekvensseillä opetettu FINPROTAX-tunnistin on tarkoitettu käytettäväksi.
Referenssit
- Roslin, T., [95 kanssakirjoittajaa] & Mutanen, M. 2021. A molecular-based identification resource for the arthropods of Finland. Molecular Ecology Resources https://doi.org/10.1111/1755-0998.13510.
- Somervuo P, Koskela S, Pennanen J, Nilsson HR, and Ovaskainen O. Unbiased probabilistic taxonomic classification for DNA barcoding. Bioinformatics. 2016 Oct 1;32(19):2920-7. doi: 10.1093/bioinformatics/btw346
- Somervuo P, Yu DW, Xu CCY, Ji Y, Hultman J, Wirta H, and Ovaskainen O. Quantifying uncertainty of taxonomic placement in DNA barcoding and metabarcoding. Methods in Ecology and Evolution. 2017 8,(4): 398-407. doi: 10.1111/2041-210X.12721